Pool-Seq
Zeitreihenstudie von Lumbricus terrestris
Im Rahmen von TrendDNA nutzen wir die langfristigen Probenahmen der Umweltprobenbank des Bundes (UPB) an mehreren Standorten in Deutschland über die letzten drei Jahrzehnte. In diesem Projekt analysieren wir Homogenatproben des weit verbreiteten Regenwurms Lumbricus terrestris, die von 1990 bis 2021 an den meisten Standorten jährlich gesammelt wurden. Dieses umfangreiche Probenahmeprogramm bietet die einzigartige Möglichkeit, signifikante selektive Veränderungen in den Regenwurmgenomen über diesen umfassenden Zeitraum nachzuweisen und möglicherweise die Ursachen für die beobachteten genomischen Veränderungen zu ermitteln. Dazu ist zunächst ein Referenzgenom für die Art erforderlich.
Aufbau des Genoms
Wir assemblieren zunächst das Regenwurmgenom eines Individuums, das im Frühjahr 2022 im Nidda-Park (Frankfurt am Main) gesammelt wurde. Nach der korrekten Identifizierung des Individuums durch taxonomische Experten und Barcoding-Ansätze verwenden wir Next-Generation Sequencing (NGS), einen parallelen Sequenzierungsansatz, der es ermöglicht, ganze Genome mit sehr hoher Genauigkeit zu erfassen. Nach der Sequenzierung verwenden wir modernste bioinformatische Softwaretools, um das vollständige Regenwurmgenom bis auf Chromosomenebene zu ermitteln. Wir verwenden das assemblierte Genom als Referenzgenom für die Zeitreihenanalysen.
Zeitreihenanalysen
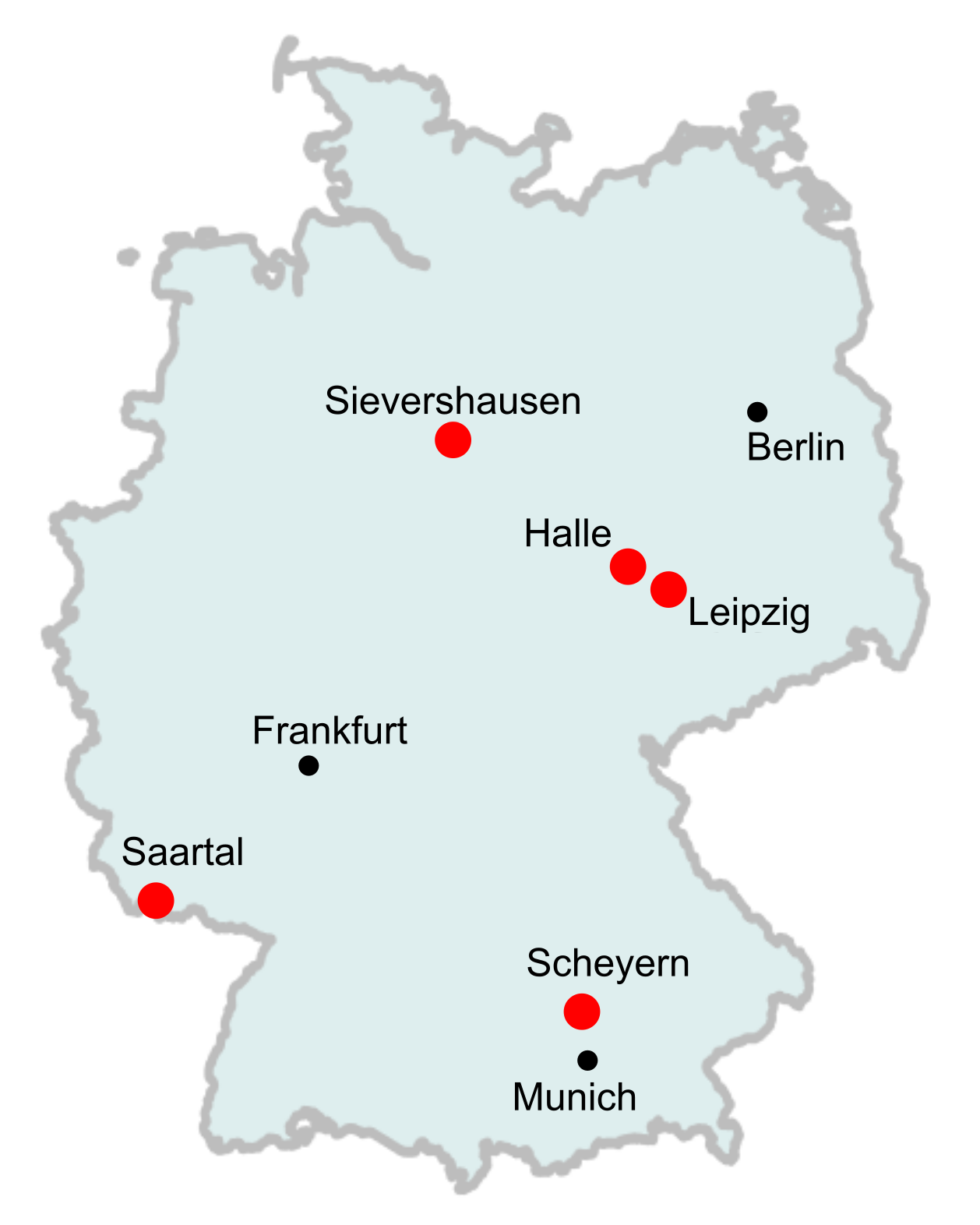
Wir verwenden fünf UPB-Langzeitprobenstandorte: Leipzig, Halle, Saartal, Scheyern und Sievershausen (Abbildung 1). Diese Standorte decken einen breiten und variablen geografischen Bereich ab, der eine Vielzahl unterschiedlicher Umweltbedingungen in ganz Deutschland abbildet. An jedem Standort wurden im Herbst in regelmäßigen Abständen Proben entnommen und bei -80 °C gelagert. Anstatt jeden Wurm einzeln zu sequenzieren, was sehr kostspielig und arbeitsintensiv wäre, fassen wir viele Individuen zusammen und stellen sicher, dass jedes Individuum ungefähr die gleiche Menge an Gewebe zum Pool beiträgt. Aufgrund dieses gleichen Beitrags sequenzieren wir jede genetische Variante entsprechend ihrer tatsächlichen Häufigkeit in der Population. Durch das Pooling von Proben erhalten wir so genaue Schätzungen zahlreicher genomischer Parameter der Population. Insgesamt umfasst diese Studie 90 Proben an den verschiedenen Standorten, wobei jede Probe bis zu 100 homogenisierte Individuen enthält. Das Ausmaß der Veränderungen im Laufe der Zeit wird uns Aufschluss darüber geben, ob in den jeweiligen Populationen eine evolutionäre Selektion stattgefunden hat.