Tagung und Workshops zu genetischen Methoden in der Umweltbeobachtung, 26-28.03.2025, Essen
Im Jahr 2025 wird das TrendDNA Projekt sich an einer dreitägigen DACHLiLux Tagung zum Thema ‚genetische Methoden in der Umweltbeobachtung‘ beteiligen. Der dritte Tag der Tagung (28.03.2025) legt den Fokus auf die Arbeiten der Umweltprobenbank des Bundes, welche im Rahmen des TrendDNA Projektes durchgeführt wurden. Wir möchten alle Interessenten auf die offizielle Seite des Umweltbundesamtes hinweisen, auf der Sie sich für die Tagung anmelden können. Der untenstehende Link wird Sie weiterleiten.
Wir hoffen Sie dort begrüßen zu können und unsere Erkenntnisse zu teilen und zu diskutieren.
https://www.umweltbundesamt.de/tagung-workshops-zu-molekularen-methoden-in-der
Mai 2024
Die IUCN veröffentlichte eine Stellungnahme zum Nutzen von eDNA Methoden im Rahmen von Konservation und Wildtier Schutzmaßnahmen. Diese Stellungnahme ist ein weiteres Signal hinsichtlich der wachsenden Bedeutung von Umwelt DNA als nicht-invasive und effektive Methode zur Beobachtung der Biologischen Vielfalt.

25. Mai 2024
Florian Leese stellte den Status Quo und Visionen zum Nutzen von DNA und eDNA basierten Methoden im Rahmen der Observation von Biodiversität als Keynote Präsentation auf dem 5. Nationalen Symposium zu eDNA basierten biologischen Begutachtungen in Shenzhen, China.


Am 11. April 2024 hielt Florian Leese eine Präsentation mit dem Titel „Biodiversität – oder das Ende der Vielfalt?“. Die Präsentation war Teil eines Sience Slam unter dem Thema „Nachhaltigkeit“ in Mainz.


November 2023, Fraunhofer IME
WildOMICs wird im Newsletter des Fraunhofer IME vorgestellt.Die Initiative hat das Ziel OMIC Technologien in Umwelt Beobachtung einzugliedern, wie etwas Meta-Transkriptomik.

September 2023
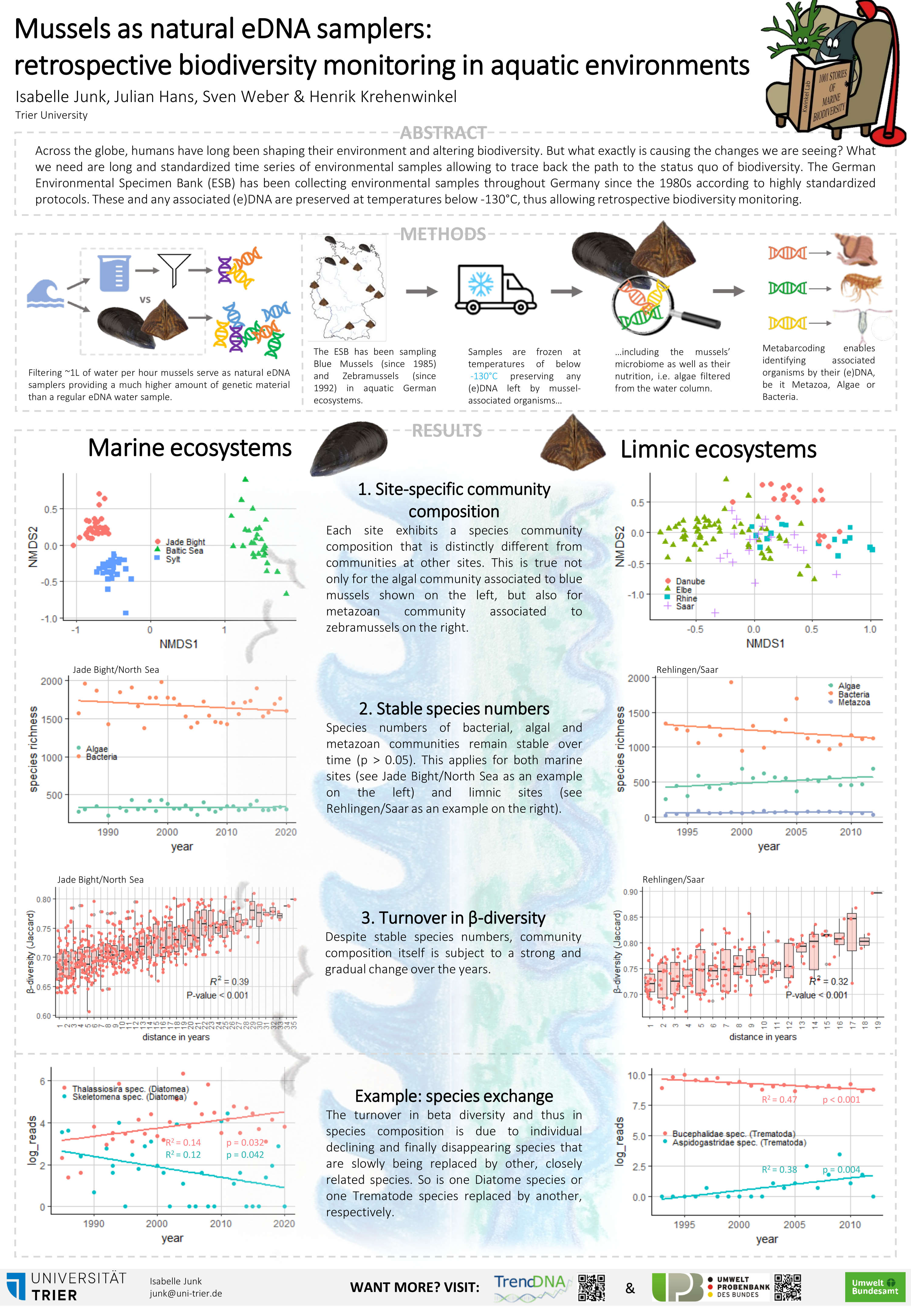
Isabelle Junk, Nina Schmitt und Henrik Krehenwinkel haben einen wissenschaftlichen Artikel Im Journal Current Biology veröffentlicht. Die Mitglieder von TrendDNA testeten erfolgreich die Miesmuschel Homogenate des Deutschen UPB-Projektes auf die Eigenschaften Umwelt DNA der Gemeinschaften um die Muscheln zum Zeitpunkt der Probennahme festzuhalten. Die Autoren konnten unter anderem den Verlauf der Invasion einer Seepocke rekonstruieren.
18. Juni 2023
An der neuen Mündung der Emscher, dem einst dreckigsten Fluss Deutschlands, fand der 1. Tag der lebendigen Emscher statt. Zu dem von Emschergenossenschaft und NABU NRW organisierten Tag trafen sich Forscher:innen, um die Artenvielfalt in und um die Emscher im Bereich der neuen Mündung zu erfassen. Auch das TrendDNA-Team der Universität Duisburg-Essen war vertreten. Robin Schütz und Florian Leese sowie Till-Hendrik Macher (GeDNA-Projekt) analysierten vor Ort mit einem portablen Sequenzierer Spuren tierischer Arten über aus dem Wasser extrahierte Umwelt-DNA. Insbesondere zahlreiche Fischarten, darunter auch der Aal und der Neunstachlige Stichling, konnten mit Hilfe der Umwelt-DNA nachgewiesen werden.

24.-25. April 2023
Mitglieder des TrendDNA Projektes Jan Koschorreck (UBA), Henrik Krehenwinkel, Julian Hans und Isa Junk (Universität Trier), Robin Schütz (Universität Duisburg-Essen), und Bernd Göckener (Fraunhofer IME) präsentieren einen Fortschrittsbericht und Aussichten des Projektes auf der UPB Jahrestagung in Berlin.

13.-15. März 2023
Aufregende neue Daten des TrendDNA Projektes wurden auf der 3. Global Soil Biodiversity Conference in Dublin (Irland) präsentiert. Im Bild rechts präsentiert Master Studentin Judith Paetsch (Senckenberg Gesellschaft, LOEWE-Zentrum-TBG), die neuesten Ergebnisse Ihrer Abschlussarbeit. Mit Hilfe von Shotgun Metagenomics charakterisierte Sie die Biodiversität der Bodengemeinschaften über räumliche und zeitliche Verteilungen.
Dezember 2022
Unsere Studie über eDNA basiertes Arthropoden Biomonitoring von Laub ist ab sofort frei zugänglich auf eLife! Diese innovative Studie wurde geleitet von Prof. Henrik Krehenwinkel von der Universität Trier. Man nutzte UPB Proben um Trends in Diversität und relativer Biomasse von Baumkronendach bewohnenden Arthropoden über die letzten 30 Jahre zu messen. Während der Artenreichtum an individuellen Standorten relativ stabil blieb, änderte sich die Artzusammensetzung drastisch. Dies führte zu einer allseitigen Homogenisierung der Wald-Arthropoden-Gesellschaften in ganz Deutschland.
2.-3. November 2022
TrendDNA Mitglieder versammelten sich am Fraunhofer IME für eine Diskussion, die zum Nachdenken über mögliche zukünftige Forschungsrichtungen anregen soll und einer Führung durch die Probenbank:



27.-28. September 2022, Universität Duisburg-Essen
Die Doktoranden Robin Schütz (Universität Duisburg-Essen) und Isabelle Junk (Universität Trier) präsentierten Poster mit Ihren spannenden Forschungen über das Potenzial von suspendierten Feststoffteilchen und Muscheln, Änderungen in limnischen und marinen Gemeinschaften aufzudecken.
Sommer 2022
Ein Begleitprojekt zu TrendDNA wurde durch die Bauer-Stiftung zur Förderung von Wissenschaft und Forschung finanziert. Unter der Führung von Dr. Susan Kennedy, Prof. Henrik Krehenwinkel und Prof. Christoph Emmerling (Universität Trier), und in Kollaboration mit Prof. Florian Leese (Universität Duisburg Essen), Prof. Miklós Bálint (Senckenberg TBG Frankfurt) und Prof. Thomas Udelhoven (Universität Trier) sollen im Rahmen dieses spannenden Projektes Metabarcoding, Metagenomik und Metatranskriptomik von Regenwürmern, Änderungen in Bodengemeinschaften über zeitliche und räumliche Verteilungen aufdecken.
Weitere Updates finden Sie auf unserer Twitter-Seite!